Picoplancton photosynthétique
Le picoplancton photosynthétique (aussi appelé picophytoplancton) est la fraction du phytoplancton dont la taille est comprise entre 0,2 et 2 µm (picoplancton).
Pages similaires :
- exclusifs précédemment cités (Prochlorococcus et Synechococcus), le troisième groupe du. picoplancton photosynthétique est composé d'eucaryotes.... (source : sb-roscoff)
Le picoplancton photosynthétique (aussi appelé picophytoplancton) est la fraction du phytoplancton dont la taille est comprise entre 0,2 et 2 µm (picoplancton). Il est particulièrement important dans les zones centrales océaniques dites oligotrophes (très pauvres en nutriments).
Historique
- 1952 : Description de la première espèce réellement picoplanctonique, Chromulina pusilla, par Butcher[1]. Cette espèce sera renommée en 1960 Micromonas pusilla[2] et est l'une des plus abondantes dans les milieux cotiers tempérés.
- 1979 : Découverte des Synechococcus marins par Waterbury[3] et confirmation par microscopie électronique par Johnson et Sieburth[4].
- 1982 : Les mêmes Johnson et Sieburth mettent aussi en lumière l'importance des petits eucaryotes par microscopie électronique[5].
- 1983 : W. K. Li et Platt mettent en évidence qu'une fraction très importante de la production primaire océanique est attribuable à des organismes d'un taille inférieure à 2 µm[6].
- 1986 : Découverte des «prochlorophytes» par Chisholm et Olson en mer des Sargasses[7], baptisés en 1992 Prochlorococcus marinus[8].
- 1995 : Découverte dans l'étang de Thau du plus petit eucaryote photosynthétique connu, Ostreococcus tauri, par Courties[9].
- 2001 : Par séquençage de l'ARN ribosomal prélevé dans des échantillons marins, plusieurs équipes européennes mettent simultanément en évidence la grande diversité du picoplancton eucaryote[10][11].
Méthodes d'étude
Sa très petite taille rend le picoplancton difficile à étudier par les méthodes classiques, dont la microscopie optique. On a par conséquent recours à d'autres méthodes ;
- Microscopie à épifluorescence. Elle permet de compter certains groupes de cellules possédant des pigments fluorescents tels que Synechococcus qui posséde phycoérythrine.
- Cytométrie en flux. Elle permet de mesurer la taille ("side scatter" en anglais) et la fluorescence sur 1 000 à 10 000 cellules par seconde, façilitant la mesure de la concentration des différentes populations picoplanctoniques sur des échantillons marins. Trois groupes de cellules (Prochlorococcus, Synechococcus et picœucaryotes) peuvent être distinguées. AinsiSynechococcus est caractérisé par la double fluorescence de ses pigments : orange pour la phycoérythrine et rouge pour la chlorophylle. La cytométrie en flux permet aussi un tri de populations spécifiques (par exemple Synechococcus) pour les cultiver ou en faire des analyses détaillées.
- Analyse des pigments photosynthétiques tels que la chlorophylle ou les caroténoïdes par chromatographie haute précision (HPLC). Elle permet de déterminer les différents groupes d'algues d'un échantillon.
- Biologie moléculaire :
-
- Clonage et séquençage de gènes tel que celui de l'ARN ribosomal. Ils permettent de déterminer la diversité totale d'un échantillon.
- DGGE (Denaturing Gradient Gel Electrophoresis), plus rapide que la technique précédente, il donne une idée de la diversité globale d'un échantillon.
- Hybridation in situ (FISH) Elle utilise des sondes fluorescentes reconnaissant des taxons précis (espèce, genre ou classe... [12].
- PCR en temps réel Elle permet de déterminer (comme le FISH) l'abondance de groupes précis et d'analyser rapidement d'un grand nombre d'échantillons[13], mais demande plus de contrôles.
Composition
Le picoplancton photosynthétique est composé de trois groupes principaux d'organismes.
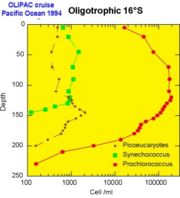
- Les cyanobactéries du genre Synechococcus d'une taille de 1 µm (micromètre) ont été les premières a être découvertes en 1979[3] mais ne dominent que dans les zones mésotrophes. Par contre, elles sont ubiquistes s'étendant des regions polaires à l'équateur.
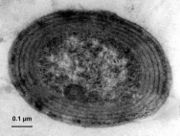
- Les cyanobactéries du genre Prochlorococcus sont particulièrement remarquables. D'une taille de 0,6 µm, Prochlorococcus n'a été découvert qu'en 1986 par deux chercheurs américains, S. W. Chisholm et R. J. Olson[7]. Malgré sa petite taille, cet organisme photosynthétique est probablement le plus abondant de la planète terre : en effet sa densité peut atteindre jusqu'à 100 millions de cellules par litre et on peut le rencontrer jusqu'à 150 m de profondeur dans toute la ceinture intertropicale[14].
- Les eucaryotes picoplanctoniques sont les moins bien connus, comme le démontre la découverte récente de groupes majeurs. Andersen et al. ont créé en 1993 une nouvelle classe d'algues brunes, celle des pelagophycées [15]. Plus surprenante encore, la découverte en 1994 d'un picœucaryote de très petite taille, Ostreococcus tauri[9], dominant la biomasse phytoplanctonique de l'étang de Thau, montre que ces organismes peuvent aussi jouer un rôle écologique important en milieu côtier ou lagunaire. En 1999, une nouvelle classe d'algue a été découverte, les bolidophycées [16], très proche génétiquement des diatomées, mais ne leur ressemblant pas du tout. A présent une cinquantaine d'espèces ont été décrites.
-
-
-
Classes d'algues contenant des espèces picoplanctoniques Classes Genres picoplanctoniques Chlorophycées Nannochloris Prasinophycées Micromonas, Ostreococcus, Pycnococcus Prymnesiophycées Imantonia Pélagophycées Pelagomonas Bolidophycées Bolidomonas Dictyochophycées Florenciella
-
-
Diversité naturelle
L'introduction de la biologie moléculaire en océanographie a révolutionné la connaissance des écosystèmes océaniques. Pour la première fois, on a pu déterminer la composition du compartiment picoplanctonique sans avoir ni à l'observer, ni à le cultiver. En pratique, on détermine la séquence d'un gène présent dans tous les organismes vivants, celui codant la petite sous-unité de l'ARN ribosomal (ARNr). Chaque espèce possédant un séquence qui lui est propre et deux espèces proches (par exemple, l'homme et le chimpanzé) ayant des séquences très semblables, l'analyse de sa séquence permet de placer un organisme dans l'arbre phylogénétique du vivant. De plus on peut déterminer sur ce gène de petites régions caractéristiques d'un groupe d'organismes (par exemple un genre du picoplancton comme Ostreococcus) et synthétiser une «sonde» reconnaissant cette région. Si cette sonde est marquée avec un composé fluorescent et mis en contact avec des cellules, seules les cellules appartenant au groupe ciblé seront visibles sous un microscope à fluorescence (technique de FISH) (voir Méthodes d'étude).
Ces approches mises en œuvre dès les années 1990 pour les bactéries, n'ont été appliquées aux picœucaryotes photosynthétiques que 10 ans plus tard. Elles ont révélées un très grande diversité[10][11] et mis en lumière l'importance des groupes suivants dans le picoplancton :
- Prasinophyceæ
- Haptophyta
- Cryptophyta
En milieu cotier tempéré, le genre Micromonas (Prasinophyceæ) semble dominant. Cependant, dans de nombreux milieux océaniques, les espèces dominantes de picoplancton eucaryote demeurent encore inconnues.
Ecologie
Chaque population picoplanctonique occupe une niche écologique différente dans le milieu océanique. Les populations de picoplancton semblent régulées non seulement par la prédation, mais également par de très virus spécifiques qu'on débute uniquement à découvrir, et que certains classent dans le nanoplancton et plus exactement dans le Femtoplancton (<0,2 µm), bien qu'ils ne soient généralement pas considérés comme des organismes vivant.
- La cyanobactérie Synechococcus est en général abondante dans les milieux mésotrophes par exemple au voisinage de l'upwelling equatorial ou en milieu côtier
- Prochlorococcus remplace Synechococcus dès que le milieu s'appauvrit (c. a. d. devient oligotrophe). Par contre en région tempérée (par exemple dans l'Atlantique Nord), Prochlorococcus est absent car les eaux trop froids ne facilitent pas son développement.
- Quant aux eucaryotes, à leur diversité taxonomique encore mal connue, correspond probablement une grande diversité d'habitats. En milieu océanique, on les trouve cependant fréquemment en profondeur à la base de la couche éclairée (couche euphotique). En milieu côtier, certaines espèces de picœucaryotes tels que Micromonas dominent. Leur abondance suit un cycle saisonnier, comme le plancton de plus grande taille, avec un maximum en été.
Il y a une trentaine d'année on faisait l'hypothèse que la vitesse de division des micro-organismes dans les écosystèmes océaniques centraux était très faible, de l'ordre de la semaine ou du mois. Cette hypothèse était confortée par le fait que la biomasse (estimée par exemple par le contenu en chlorophylle) était très stable au cours du temps. Toutefois avec la découverte du picoplancton, on s'est aperçu que le système était bien plus dynamique qu'il n'y paraissait. En spécifique, on a découvert l'ubiquité de minuscules prédateurs d'une taille de quelques microns qui ingéraient les algues picoplanctoniques aussi rapidement qu'elles étaient produites. Ce système prédateur-proie extrêmement sophistiqué est quasiment systématiquement à l'équilibre et résulte en une biomasse quasi-constante. Cette idéale adéquation entre production et consommation rend cependant extrêmement difficile la mesure précise de la vitesse du système.
En 1988, deux chercheurs américains, Carpenter et Chang[17] avaient proposé d'estimer la vitesse de division des cellules du phytoplancton à partir du suivi de la réplication de l'ADN par microscopie. En remplaçant la microscopie par la cytométrie en flux, il est possible suivre le contenu en ADN des cellules de picoplancton (par exemple Prochlorococcus) au cours du temps. On s'est dans ce cas aperçu que les cellules du picoplancton sont extrêmement synchrones[18] : elles répliquent leur ADN puis se divisent toutes en même temps en fin de journée. Cette synchronisation pourrait être due à la présence d'une horloge cellulaire interne.
Le picoplancton occupe une place encore mal évaluée dans les réseaux trophiques.
Génomique
Dans les années 2000, la génomique a permis de franchir une étape supplémentaire. La génomique consiste à déterminer la séquence complète du génome d'un organisme ainsi qu'à répertorier tous les gènes présents. Ainsi on peut avoir une idée des capacités métaboliques de l'organisme ciblé et comprendre comment il s'adapte à son environnement. A ce jour, les génomes de plusieurs souches de Prochlorococcus[19][20] et de Synechococcus[21], et d'une souche d'Ostreococcus [22] ont été déterminés, alors que ceux de plusieurs autres cyanobactéries et de petits eucaryotes (Bathycoccus, Micromonas) sont en cours d'analyse. En parallèle, on débute aussi à faire des analyses génomiques directement à partir d'échantillons océaniques (écogénomique ou métagénomique) [23], ce qui permet d'accèder aux génomes d'organismes non-cultivés.
-
-
-
Génomes séquencés de souches de picoplancton photosynthétique Genre Souche Centre de séquencage Référence Prochlorococcus MED4 JGI SS120 Genoscope MIT9312 JGI MIT9313 JGI NATL2A JGI CC9605 JGI CC9901 JGI Synechococcus WH8102 JGI WH7803 Genoscope RCC307 Genoscope CC9311 TIGR [24] Ostreococcus OTTH95 Genoscope
-
-
Voir aussi
Références
Références citées
- ↑ Butcher, R. (1952). Contributions to our knowledge of the smaller marine algæ. Journal of the Marine Biological Association U. K. 31 : 175-91.
- ↑ Manton, I. & Parke, M. (1960). Further observations on small green flagellates with special reference to possible relatives of Chromulina pusilla Butcher. Journal of the Marine Biological Association U. K. 39 : 275-98.
- ↑ a b Waterbury, J. B. et al. (1979). Wide-spread occurrence of a unicellular, marine planktonic, cyanobacterium. Nature 277 : 293-4.
- ↑ Johnson, P. W. & Sieburth, J. M. (1979). Chroococcoid cyanobacteria in the sea : a ubiquitous and diverse phototrophic biomass. Limnology and Oceanography 24 : 928-35.
- ↑ Johnson, P. W. & Sieburth, J. M. (1982). In-situ morphology and occurrence of eucaryotic phototrophs of bacterial size in the picoplankton of estuarine and oceanic waters. Journal of Phycology 18 : 318-27.
- ↑ Li, W. K. W. et al. (1983). Autotrophic picoplankton in the tropical ocean. Science 219 : 292-5.
- ↑ a b Chisholm, S. W. et al. (1988). A novel free-living prochlorophyte occurs at high cell concentrations in the oceanic euphotic zone. Nature 334 : 340-3.
- ↑ Chisholm, S. W. et al. (1992). Prochlorococcus marinus nov. gen. nov. sp. : an oxyphototrophic marine prokaryote containing divinyl chlorophyll a and b. Archives of Microbiology 157 : 297-300.
- ↑ a b Courties, C. et al. (1994). Smallest eukaryotic organism. Nature 370 : 255.
- ↑ a b López-García, P. et al. (2001). Unexpected diversity of small eukaryotes in deep-sea Antarctic plankton. Nature 409 : 603-7.
- ↑ a b Moon-van der Staay, S. Y. et al. (2001). Oceanic 18S rDNA sequences from picoplankton reveal unsuspected eukaryotic diversity. Nature 409 : 607-10.
- ↑ Not, F. et al. (2004). A single species Micromonas pusilla (Prasinophyceæ) dominates the eukaryotic picoplankton in the western English Channel. Applied and Environmental Microbiology 70 : 4064-72.
- ↑ Johnson, Z. I. et al. (2006). Niche partitioning among Prochlorococcus ecotypes along ocean-scale environmental gradients. Science 311 : 1737-40.
- ↑ Partensky, F. et al. (1999). Prochlorococcus, a marine photosynthetic prokaryote of global significance. Microbiology and Molecular Biology Reviews 63 : 106-27.
- ↑ Andersen, R. A. et al. (1993). Ultrastructure and 18S rRNA gene sequence for Pelagomonas calceolata gen. and sp. nov. and the description of a new algal class, the Pelagophyceæ classis nov. Journal of Phycology 29 : 701-15.
- ↑ Guillou, L. et al. (1999). Bolidomonas : a new genus with two species belonging to a new algal class, the Bolidophyceæ (Heterokonta). Journal of Phycology 35 : 368-81.
- ↑ Carpenter, E. J. & Chang, J. (1988). Species-specific phytoplankton growth rates via diel DNA synthesis cycles. I. Concept of the method. Marine Ecology - Progress Series 43 : 105-11.
- ↑ Vaulot, D. et al. (1995). Growth of Prochlorococcus a photosynthetic prokaryote, in the equatorial Pacific Ocean. Science 268 : 1480-2.
- ↑ Rocap, G. et al. (2003). Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation. Nature 424 : 1042-7.
- ↑ Dufresne, A. et al. (2003). Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome. Proceedings of the National Academy of Sciences of the United States of America 100 : 10020-5.
- ↑ Palenik, B. et al. (2003). The genome of a motile marine Synechococcus. Nature 424 : 1037-42.
- ↑ Derelle, E. et al. (2006). Genome analysis of the smallest free-living eukaryote Ostreococcus tauri unveils many unique features. Proceedings of the National Academy of Sciences of the United States of America 103 : 11647-52.
- ↑ Venter, J. C. et al. (2004). Environmental genome shotgun sequencing of the Sargasso Sea. Science 304 : 66-74.
- ↑ Palenik, B. et al. (2006). Genome sequence of Synechococcus CC9311 : Insights into adaptation to a coastal environment. PNAS 103 : 13555-9.
Autres références
Cyanobactéries
- Zehr, J. P., Waterbury, J. B., Turner, P. J., Montoya, J. P., Omoregie, E., Steward, G. F., Hansen, A. & Karl, D. M. 2001. Unicellular cyanobacteria fix N2 in the subtropical North Pacific Ocean. Nature 412 :635-8
Eucaryotes
- Eikrem, W., Throndsen, J. 1990. The ultrastructure of Bathycoccus gen. nov. and B. prasinos sp. nov., a non-motile picoplanktonic alga (Chlorophyta, Prasinophyceæ) from the Mediterranean and Atlantic. Phycologia 29 :344-350
- Chrétiennot-Dinet, M. J., Courties, C., Vaquer, A., Neveux, J., Claustre, H., et al. 1995. A new marine picœucaryote : Ostreococcus tauri gen et sp nov (Chlorophyta, Prasinophyceæ). Phycologia 34 :285-292
- Sieburth, J. M., M. D. Keller, P. W. Johnson, and S. M. Myklestad. 1999. Widespread occurrence of the oceanic ultraplankter, Prasinococcus capsulatus (Prasinophyceæ), the diagnostic Golgi-decapore complex and the newly described polysaccharide capsulan'. J. Phycol. 35 : 1032-1043.
Ecologie
- Platt, T., Subba-Rao, D. V. & Irwin, B. 1983. Photosynthesis of picoplankton in the oligotrophic ocean. Nature 300 :701-4.
- Campbell, L., Nolla, H. A. & Vaulot, D. 1994. The importance of Prochlorococcus to community structure in the central North Pacific Ocean. Limnol. Oceanogr. 39 :954-61.
Biologie moléculaire et Génomes
- Rappé, M. S., P. F. Kemp, and S. J. Giovannoni. 1995. Chromophyte plastid 16S ribosomal RNA genes found in a clone library from Atlantic Ocean seawater. J. Phycol. 31 : 979-988.
Recherche sur Google Images : |
|
"Picoplancton photosynthétique - ..." L'image ci-contre est extraite du site fr.wikipedia.org Il est possible que cette image soit réduite par rapport à l'originale. Elle est peut-être protégée par des droits d'auteur. Voir l'image en taille réelle (180 x 136 - 6 ko - jpg)Refaire la recherche sur Google Images |
Recherche sur Amazone (livres) : Refaire la recherche Refaire la recherche |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 13/11/2008.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.



 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité